北京大学汤富酬团队最新Nature子刊
 2025-07-07
2025-07-07
 7050
7050

编者按
2025年7月4日,北京大学汤富酬团队在Nature Structural & Molecular Biology(IF=10.1)在线发表题为“Single-cell long-read Hi-C, scNanoHi-C2, details 3D genome reorganization in embryonic-stage germ cells”的研究论文,该研究提出了一种单细胞输入、长读Hi-C方法,称为scNanoHi-C2。

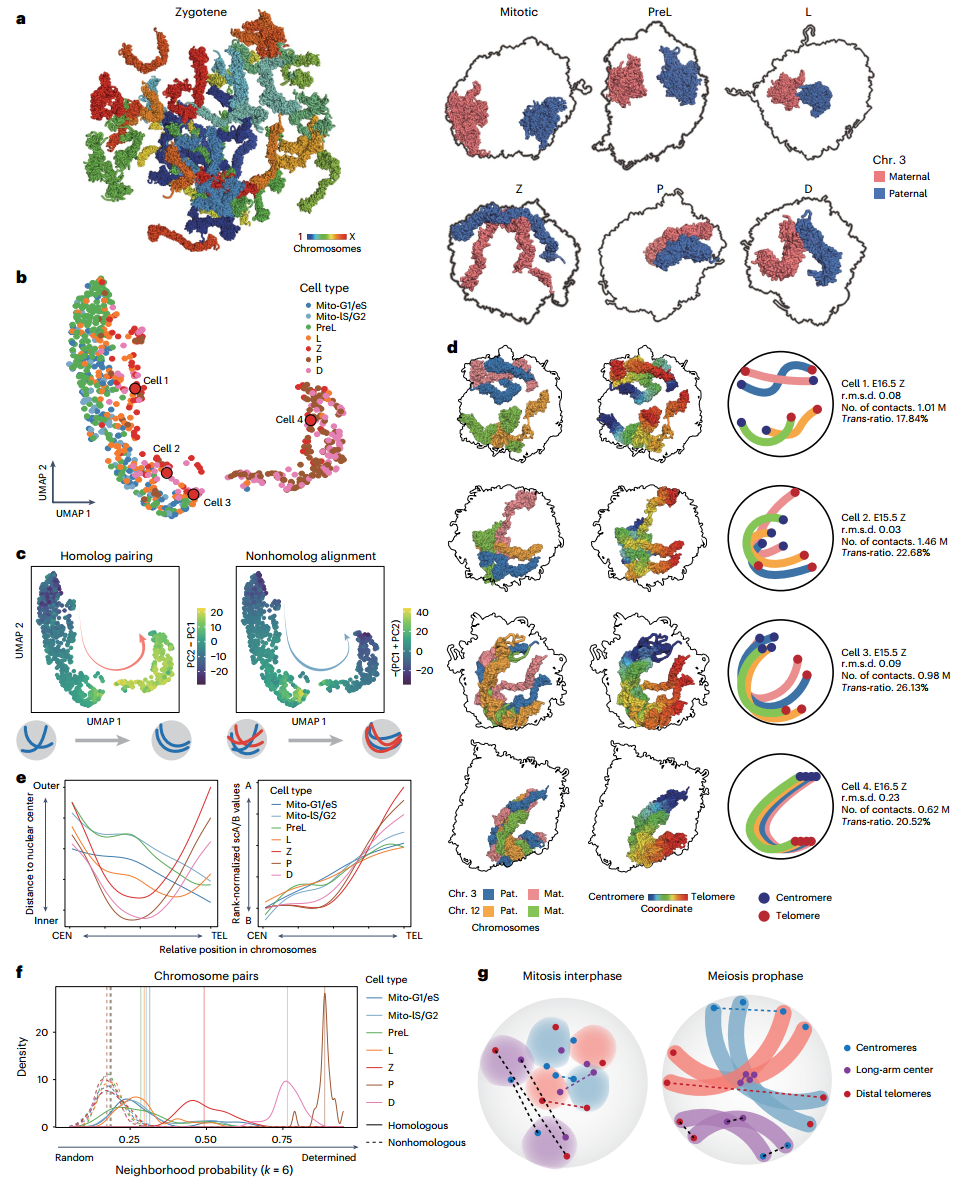
高阶相互作用协调精细的隔室,促进雌性胚胎干细胞的减数分裂程序
(图源自Nature Structural & Molecular Biology)
该研究开发了一种基于牛津纳米孔技术(ONT)平台的单细胞输入scHi-C方法,称为scNanoHi-C2,并将其应用于研究小鼠EGCs的3D基因组结构。使用scNanoHi-C2系统地剖析了EGC染色质结构的动力学。研究发现,尽管常染色体的变化与精子发生相似,但雌性EGCs的X染色体显示出B区室之间增强的特异性相互作用。通过重建3D基因组模型,观察到减数分裂过程中染色体的动态定位,表明EGCs非同源染色体之间的邻域是相对随机的。
同时,转座因子经历了戏剧性的染色质重组,并在减数分裂拓扑相关结构域边界周围显示出Alu/B2元件的不对称分布。此外,在有丝分裂阶段,EGCs中的高阶相互作用主要富集在B区室中,而在有丝分化到减数分裂过渡后,富集的高阶交互作用转移到精细的A区室,在全球基因组浓缩过程中可能促进减数分裂特异性转录程序。还揭示了有丝分裂停滞的雄性EGCs中一种意想不到的染色质结构,与之前假设的G0状态不同,这可能为随后的精子发生奠定了独特的基因组结构。总之,该研究强调了scNanoHi-C2的潜力,并揭示了EGCs染色质结构重编程的关键特征,这些结果可能有助于理解减数分裂过程中的机制和性别二态性。