顶刊再+1!看浙大“女娲”AI模型
 2025-07-10
2025-07-10
 8194
8194

iNature
2025年7月8日,浙江大学郭国骥、韩晓平共同通讯在Cell在线发表题为“Modeling the vertebrate regulatory sequence landscape by UUATAC-seq and deep learning”的研究论文,该研究开发了一种使用测序(UUATAC-seq)协议对转座酶可及染色质进行超通量、超灵敏的单核分析,该协议能够在1天的实验中为一个物种构建染色质可及性景观。
使用UUATAC-seq,绘制了五种代表性脊椎动物的候选顺式调控元件(cCRE)图谱。分析表明,不同物种的基因组大小差异会影响cCRE的数量,但不会影响其大小。研究人员引入了Nvwa顺式调控元件(NvwaCE,“女娲”),这是一个大型任务深度学习模型,旨在解释顺式调控语法,并直接从基因组序列中高精度地预测cCRE景观。NvwaCE证明了调控语法比核苷酸序列更保守,并且这种语法将cCRE组织成不同的功能模块。此外,NvwaCE准确预测了合成突变对谱系特异性cCRE功能的影响,与因果数量性状位点(QTL)和基因组编辑结果一致。总之,该研究为解码脊椎动物的调节语言提供了宝贵的资源。

单细胞技术的进步极大地增强了研究复杂生物系统中基因调控的能力。基于液滴、微孔或分流池策略的高通量单细胞测序方法,能够大规模并行分析单个细胞,而不会对细胞群体或标记列表进行有偏见的选择。这些技术革命为生物学中基因组、转录组和蛋白质组信息的系统测量设定了新的标准。生物规模细胞图谱的最新进展为细胞谱系规格和遗传调控提供了前所未有的见解。虽然转录组图谱侧重于谱系特异性基因表达,但单细胞染色质可及性图谱可以捕捉控制每种细胞类型转录的染色质调控景观。然而,目前的细胞图谱技术有多种局限性:难以平衡通量和灵敏度,DNA片段或转录物的缺失,以及使数据整合复杂化的批量效应。
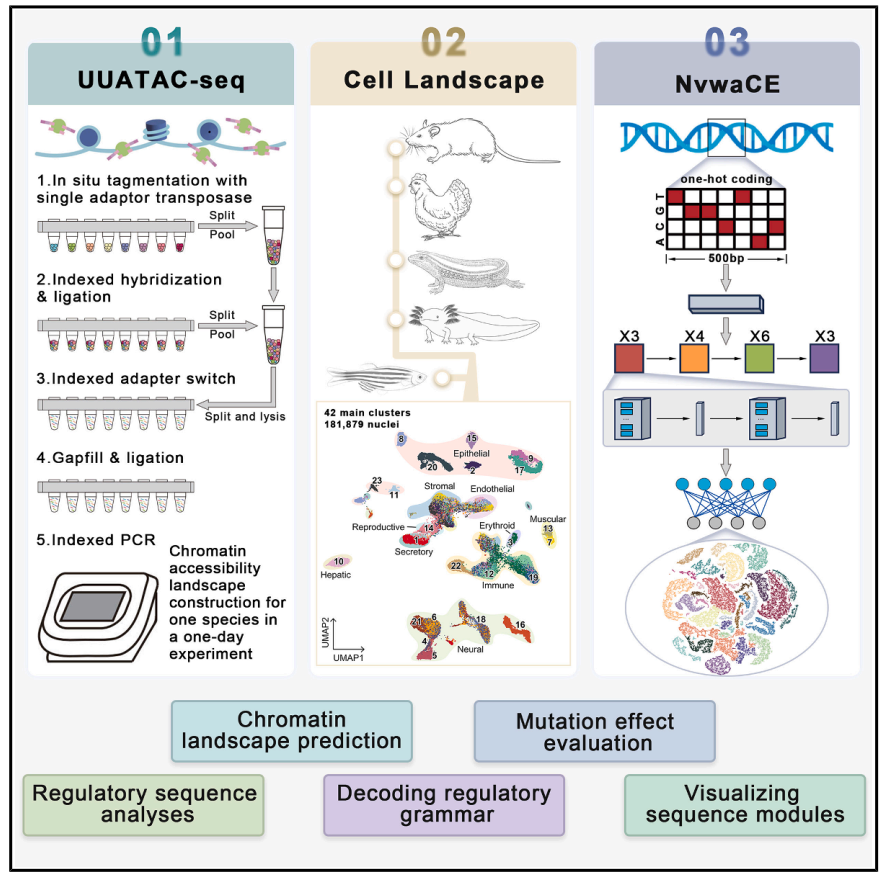
在这项研究中,研究人员通过使用同源衔接子转座酶和温控衔接子转换,显著提高了ATAC分析的灵敏度和通量。此外,还引入了一种高通量、超灵敏的单核ATAC序列(UUATAC-seq)方案来绘制斑马鱼、蝾螈、壁虎、鸡和小鼠的高分辨率染色质图谱,其基因组大小跨越了脊椎动物亚门,这使得能够研究不同物种和谱系中脊椎动物候选顺式调控元件(cCREs)的共同模式。最后,建立了一个巨型任务深度学习模型,Nvwa顺式调控元件(NvwaCE),它可以准确预测染色质可及性景观,推断进化效应,并直接从任何给定的脊椎动物基因组序列中评估合成突变。总的来说,该研究开发了能够高效构建染色质可及性图谱的UUATAC-seq技术,以及用于脊椎动物染色质图谱预测的基因组 AI 模型—女娲CE(NvwaCE),这些成果为进一步破译脊椎动物基因组的调控语言提供了宝贵资源。
论文并列一作包括22级博士生吴涵语、20级博士生王雪怡、21级博士生刘代媛、21级博士生傅雨婷、20级博士生杨蕾、博士后汪仁英、博士后张霈婧、22级博士生马立枫。该研究获得了国家自然科学基金重点项目与浙江省尖兵领雁项目的支持。